
CLASSIFICATION DES IRIS
Dans ce cas pratique, nous allons utiliser le jeu de données des Iris considéré aujourd'hui comme un cas d'école. Néanmoins, ce jeu de données va nous permettre de mettre en pratique quelques fonctions liées à l'analyse de données en utilisant le module Pandas et la réalisation d'apprentissages à l'aide d'algorithmes de machine learning dédiés à la classification.
Enfin, nous terminerons par l'utilisation de réseau de neurones (deep learning à l'aide des frameworks Tensorflow et Keras
Pour toute remarque ou question, n'hésitez pas à nous contacter, nous nous ferons un plaisir de vous répondre !
Définition du problème</H2>
En tant que responsable botanique d’un grand château situé à Versailles, j’aimerais pouvoir disposer d’un logiciel capable d’identifier quelle espèce d’iris à élue domicile dans les jardins du château.

1ère partie : Compréhension des données
Acquisition des données
Le module SciKit-Learn comporte le jeu de données dont nous avons besoin.
from sklearn import datasets
iris = datasets.load_iris()
print("> Données chargées")
Transformation des données en DataFrame grace au module Pandas
import pandas as pd
iris_df = pd.DataFrame(iris.data, columns = iris.feature_names)
iris_df.head(10)

Récupération des données à prédire, c'est à dire l'espèce d'Iris et stockage de celles-ci dans un DataFrame (0 = Setosa, 1 = Versicolor, 2 = Viriginica)
iris_type_df = pd.DataFrame(iris.target)
iris_type_df.head(5)
Fusion des deux DataFrame pour n'en obtenir qu'un seul qui contiendra les caractéristiques des Iris et l'espèce correspondante. Cette fusion se fait par l'ajout d'une colonne dans le premier DataFrame où seront copiées les données du Data Frame contenant les espèces
#Creation d'une colonne "species" contant le dataFrame iris_type_df
iris_df['species'] = iris_type_df
iris_df.head(5)
Analyse des données
Combiens avons nous d'observations et de caractéristiques ?
iris_df.shape
150 observations et 5 caractéristiques
Comment se nomment les caractéristiques ?
iris_df.columns
On constate que les noms des caractéristiques comportent des espaces, ce n'est pas très "coding friendly". Nous allons donc les renommer
iris_df = iris_df.rename(columns={"sepal length (cm)": "sepal_length",
"sepal width (cm)": "sepal_width",
"petal length (cm)": "petal_length",
"petal width (cm)": "petal_width",
})
iris_df.columns
Quel est le type des différentes caractéristiques ?
iris_df.dtypes
Toutes les caractéristiques sont de type numériques
Avons nous des données manquantes ?
iris_df.isnull().sum()
Aucune donnée manquante
Combien avons nous d'iris de chaque espèce ?(0 = Setosa, 1 = Versicolor, 2 = Viriginica) Les données sont-elles équilibrées ?
print(iris_df["species"].value_counts())
Nous disposons de 150 observations dont la répartition des espèces est équilibrée (3 espèces comportant chacune 50 observations)
iris_df.loc[:49]
iris_df.loc[50:99]
iris_df.loc[100:149]
Les données ne sont pas mélangées (les iris sont classés par espèce) ce qui peut géner l'apprentissage. Nous allons donc corriger ce point
iris_df = iris_df.sample(frac=1).reset_index(drop=True)
print(iris_df.head(10))
Visualisation de la corrélation des données entre elles
import matplotlib.pyplot as plt
import seaborn as sns
plt.figure(figsize=(15,6))
g = sns.heatmap(iris_df.corr(),
vmin=-1,
vmax=1,
cmap='coolwarm',
annot=True,
linewidths=2,
square=True);
g.set_yticklabels(g.get_yticklabels(), rotation = 0, fontsize = 8)
plt.show()
On note une forte correlation entre la caractéristique à prédire (species) et les caractéristique des pétales
Visualisation des espèces en fonction de leur sépales
import warnings
warnings.filterwarnings("ignore")
sns.set_style("whitegrid");
sns.FacetGrid(iris_df, hue="species", size=4) \
.map(plt.scatter, "sepal_length", "sepal_width") \
.add_legend();
plt.show()
On constate une catégorisation possible pour les iris de type sétosa. Cependant cela reste plus complexe pour les iris virginica et versicolor
Visualisation des espèces en fonction de leur pétales
import seaborn as sns
sns.set_style("whitegrid");
sns.FacetGrid(iris_df, hue="species", size=4) \
.map(plt.scatter, "petal_length", "petal_width") \
.add_legend();
plt.show()
Cette fois ci la catégorisation des différentes espèces d'iris est plus marquée à l'aide des caractéristiques des pétales comme nous l'indiquait le graphique des corrélations
2ème partie : Machine learning
Recherche d'un modèle
Nous sommes en présence d'un apprentissage supervisé répondant un à problème de classification. Dans ce cas, nous pouvons utiliser les modèles suivants:
- La Regression logistique
- Machine vecteurs de support (SVM)
- KNN (K plus proches voisins)
- L'arbre de décision
- Les rorêts aléatoires
- Naive Bayes
Nous allons donc créer un tableau chargé de contenir les différents algorithmes à tester
from sklearn.linear_model import LogisticRegression
from sklearn.svm import SVC
from sklearn.neighbors import KNeighborsClassifier
from sklearn.tree import DecisionTreeClassifier
from sklearn.ensemble import RandomForestClassifier
from sklearn.naive_bayes import GaussianNB
algorithmes = []
algorithmes.append(('Regression Logistique', LogisticRegression()))
algorithmes.append(('Machine vecteurs de support', SVC()))
algorithmes.append(('KNN', KNeighborsClassifier()))
algorithmes.append(('Arbre de decision', DecisionTreeClassifier()))
algorithmes.append(('Forêt aléatoire',RandomForestClassifier()))
algorithmes.append(('Naive Bayes', GaussianNB()))
print("Tableau créé...")
La première étape va consister à selectionner les caractéristiques relatives à l'apprentissage (X) et celle relative à la prédiction (Y)
X_APPRENTISSAGE = iris_df[['sepal_length','sepal_width','petal_length','petal_width']]
Y_APPRENTISSAGE = iris_df[['species']]
print("Découpage effectué...")
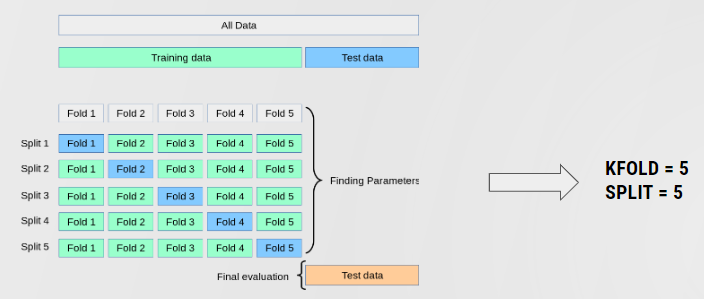
Nous pouvons tester les algorithmes les uns aprés les autres, cependant pour nous faciliter cette tache nous allons utiliser un procédé appelé cross_validation.
La première étape consiste à découper nos données (X et Y) en petits groupes (fold) qui serviront de base d'apprentissage et de test. Ce découpage est réalisé à l'aide de la fonction KFold. Dans notre cas, nous allons découper nos données en 4 petits groupes.
Voici comment la cross validation s'opère pour un algorithme :
- Le premier groupe sert de groupe de test
- Les groupes 2,3,4 servent pour l'apprentissage
- L'algorithme apprend sur les 3 groupes d'apprentissage
- Une validation de cet apprentissage est fait sur le groupe de test
- Une précision de l'apprentissage est réalisé
- Le second groupe sert de groupe de test
- Les groupes 1,3,4 servent pour l'apprentissage
- L'algorithme apprend sur les 3 groupes d'apprentissage
- Une validation de cet apprentissage est fait sur le groupe de test
Une précision de l'apprentissage est réalisé
etc...
A l'issu des différents apprentissages, une moyenne des précisions est calculé donnant alors la précision de l'algorithme.
pour plus d'information sur la cross validation, vous pouvez consulter cette adresse : https://scikit-learn.org/stable/modules/cross_validation.html#cross-validation-and-model-selection

#On ignore les warning
import warnings
warnings.filterwarnings("ignore")
#Fonction de selection de modèle
from sklearn import model_selection
#KFOld
nombreDeGroupes = 4
nombreIterations = nombreDeGroupes
kfold = model_selection.KFold(n_splits=nombreDeGroupes, random_state=101)
#On mesure la performance des algorithmes sur leur précision
score = 'accuracy'
#On stock les différents résultats
scores = []
for nom_algorithme, algorithme in algorithmes:
kfold = model_selection.KFold(n_splits=nombreDeGroupes, random_state=101)
print("\n-- "+nom_algorithme+"--")
#Appprentissage
cross_validation_resultats = model_selection.cross_val_score(algorithme, X_APPRENTISSAGE, Y_APPRENTISSAGE, cv=nombreIterations, scoring=score)
print("Précisions de chaque apprentissage : "+str(cross_validation_resultats))
scores.append(cross_validation_resultats.mean())
print("Moyenne des précisions = "+str(cross_validation_resultats.mean()))
print("\n")
print("*******************************************")
print("Meilleur algorithme : ")
print(" - "+algorithmes[scores.index(max(scores))][0]+" avec "+str(round((scores[scores.index(max(scores))])*100,2))+ " %")
print("*******************************************")
print("")
2ème partie : Deep Learning
Architecture du réseau de neurones
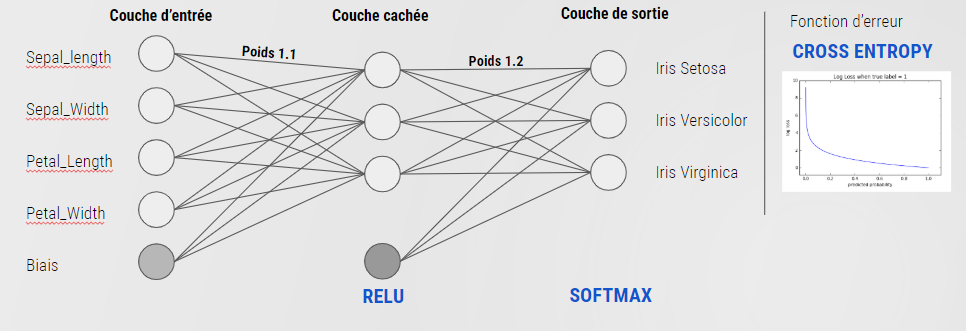
Nous avons choisi de mettre en place cette architecture :
- Nombre de neurones en entrée = Nombre de caractéristique explicatives = 4
- Nombre de neurones en sortie = Nombre de classes à prédire = 3 (0 = Setosa, 1 = Versicolor, 2 = Viriginica)
- Nombre de couche cachées = 1
- Nombre de neurones dans la couche cachée = moyenne entre le nombre de neurones en entrée et en sortie = 3
- Un biais pour la couche d'entrée
Un biais pour la couche de sortie
Pour les fonctions d'activation et d'erreur nous utiliserons (Fonctions communément utilisées pour ce type de problème) :
- Relu pour la fonction d'activation de la couche cachée
- Softmax pour la fonction d'activation de la dernière couche
- Cross Entropy pour la fonction d'erreur
Utilisation de Tensorflow
Hot encoding des espèces
Chaque observation contient un numéro d'espèce (caractéristique species) : 0 = Setosa, 1 = Versicolor, 2 = Viriginica Dans notre réseau de neurones, nous avons 3 neurones de sorties correspondant chacun à une espèce. Nous allons donc devoir traduire l'espèce de l'observation pour qu'elle soit compréhensible par notre réseau de neurones :
- Si c'est un iris setosa nous aurons [1,0,0] 1 pour le premier neurone et 0 pour les autres
- Si c'est un iris versicolor nous aurons [0,1,0] 1 pour le second neurone et 0 pour les autres
- Si c'est un iris virginica nous aurons [0,0,1] 1 pour le troisième neurone et 0 pour les autres
Pour réaliser cela, nous allons ajouter 3 colonnes à notre dataframe grâce à la méthode get_dummies du module pandas.
import numpy as np
iris_df = pd.get_dummies(iris_df, columns=['species'])
print(iris_df.head(10))
Découpage des données en jeu d'apprentissage (70% du jeu de données complet) et jeu de test (30% du jeu de données complet)
#Extraction des caractéristiques
X = iris_df[['sepal_length','sepal_width','petal_length','petal_width']]
y = iris_df [['species_0','species_1','species_2']]
#Transformation des données en tableau numpy (Pour Tensorflow)
y = np.array(y, dtype='float32')
X = np.array(X, dtype='float32')
#Decoupage en jeu de d'apprentissage et de test
#Test size = 30%
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=101)
print("Découpage effectué...")
Paramétrage du réseau de neurones
import tensorflow as tf
# Couches d'entrée et de sortie
couche_entree = tf.placeholder(shape=[None, 4], dtype=tf.float32)
couche_sortie = tf.placeholder(shape=[None, 3], dtype=tf.float32)
#Nombre de neurones sur la couche cachée
nb_neurones_couche_cachée = 3
# Poids des différentes couches
w1 = tf.Variable(tf.random_normal(shape=[4,nb_neurones_couche_cachée])) # Couche entrée -> couche cachée
w2 = tf.Variable(tf.random_normal(shape=[nb_neurones_couche_cachée,3])) # couche cachée -> couche de sortie
#Biais
b1 = tf.Variable(tf.random_normal(shape=[nb_neurones_couche_cachée])) #Biais de la couche d'entrée
b2 = tf.Variable(tf.random_normal(shape=[3])) # Biais de la couche cachée
#Fonctions d'activation :
hidden_output = tf.nn.relu(tf.add(tf.matmul(couche_entree, w1), b1)) # Relu
final_output = tf.nn.softmax(tf.add(tf.matmul(hidden_output, w2), b2)) #Softmax
print("Réseau paramétré...")
Fonction d'erreur à minimiser
Les fonction d'activation et de minimisation d'erreur utilisées dans notre réseau de neurones, sont propres à une configuration dédiée à la classification devenue "Standard" pour son obtention de bons résultats. Cette configuration comporte une activation de type softmax pour la dernière couche de neurone et une fonction d'erreur de type cross entropy. Pour plus d'informations: https://ml-cheatsheet.readthedocs.io/en/latest/loss_functions.html
#Formule de calcul de l'erreur (cross entropy)
erreur = tf.reduce_mean(-tf.reduce_sum(couche_sortie * tf.log(final_output), axis=0))
# Minimisation de l'erreur à l'aide de l'algorithme de descente de gradient comprenant
# un taux d'apprentissage de 0.001
optimiseur = tf.train.GradientDescentOptimizer(learning_rate=0.001).minimize(erreur)
print("Paramétrage effectué...")
Initialisation du réseau et apprentissage
# Initialisation des variables (poids...)
init = tf.global_variables_initializer()
#Nombre de boucles d'apprentissage
epochs = 1000
#Session tensorflow
session = tf.Session()
session.run(init)
#Création d'un tableau afin de représenter l'erreur commise lors de l'apprentissage
Graphique_Erreur=[]
# Apprentissage
print('Apprentissage du modèle...')
for i in range(1, (epochs + 1)):
#Apprentissage
session.run(optimiseur, feed_dict={couche_entree: X_train, couche_sortie: y_train})
#Calcul de l'erreur
erreur_epoch = session.run(erreur, feed_dict={couche_entree: X_train, couche_sortie: y_train})
Graphique_Erreur.append(erreur_epoch)
print('Epoch', i, '|', 'Erreur :',erreur_epoch )
#Affichage du graphique
plt.plot(Graphique_Erreur)
plt.show()
Réalisation de quelques prédictions
nbPredictions = 0
nbBonnesPredictions = 0
for i in range(len(X_test)):
nbPredictions = nbPredictions+1
#Prediction attendue
attendu = y_test[i]
#Prediction
predition = np.rint(session.run(final_output, feed_dict={couche_entree: [X_test[i]]}))
print('Reel:', y_test[i], 'Predit:', predition[0])
if (str(y_test[i]) == str(predition[0])):
nbBonnesPredictions = nbBonnesPredictions+1
print ("\nPrecision = "+str((nbBonnesPredictions/nbPredictions)*100)+" %")
Utilisation de KERAS
Configuration du réseau de neurones
from keras import models
from keras import layers
from keras.models import Sequential
from keras.layers import Dense
#Suppression des warning (3 = les messages de type INFO, WARNING et ERROR seront non affichés)
import os
os.environ['TF_CPP_MIN_LOG_LEVEL'] = '3'
#Modèle de type séquentiel (Empilement)
model = Sequential()
#Couche d'entrée
#(Nombre de neurones en sortie, nombre de neurones en entrée, nom, fonction d'activation)
model.add(Dense(3, input_dim=4, name='couche_entree',activation='relu'))
#Couche de sortie
model.add(Dense(3, activation='softmax', name='couche_sortie'))
print(model.summary())
Initialisation du réseau et apprentissage
#Suppression des warning (3 = les messages de type INFO, WARNING et ERROR seront non affichés)
import os
os.environ['TF_CPP_MIN_LOG_LEVEL'] = '3'
#On retrouve la fonction d'erreur cross_entropy
model.compile(optimizer='adam',
loss='categorical_crossentropy',
metrics=['accuracy'])
#Apprentissage
model.fit(X_train, y_train, epochs=500)
Quelques prédictions
#Realisation des prédictions
predictions = model.predict_classes(X_test)
nbPredictions = 0
nbBonnesPredictions = 0
# show the inputs and predicted outputs
for i in range(len(predictions)):
nbPredictions = nbPredictions +1
print("Attendu=%s, Attendu=%s, Predicted=%s" % (y_test[i],np.argmax(y_test[i]),predictions[i]))
if (np.argmax(y_test[i]) == predictions[i]):
nbBonnesPredictions = nbBonnesPredictions +1
print ("\nPrecision = "+str((nbBonnesPredictions/nbPredictions)*100)+" %")